휴먼스케이프 Software engineer Covy입니다.
본 포스트에서는 딥러닝으로 희귀 유전성 질환을 진단한다는 내용의 논문에 대해서 리뷰하려고 합니다. 논문의 제목은 다음과 같습니다.
“DeepGestalt-Identifying Rare Genetic Syndromes Using Deep Learning”
논문에 대한 내용을 직접 보시고 싶으신 분은 이곳을 참고하시면 좋습니다.
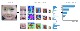
DeepGestalt: High level flow [출처: DeepGestalt-Identifying Rare Genetic Syndromes Using Deep Learning]
Objective
논문에서 목적으로 하는 바는 환자의 얼굴 사진을 이용해 그 환자의 희귀 질환을 예측해내는 딥러닝 모델을 구현하는 것입니다.
논문에서 인용한 자료에 따르면 많은 유전성 질환들이 얼굴로 표현되는 표현형(facial phenotype)을 가지기에 가능한 방법이며, 얼굴 사진은 유전학자들이 유전병을 진단하는데 실제로 도움을 주고 있는 데이터라고 합니다.
이러한 기반과 목적성을 가지고 논문에서 구체적으로 구현하고자 하는 blackbox model은 환자의 얼굴 사진 x를 input으로 넣어 similarity score(논문에서는 이를 Gestalt score라고 정의합니다)가 포함된 유전성 질환의 sorted list를 output으로 산출하는 함수 f(x)입니다.
Methods
Image preprocessing
가장 첫 단계는 image preprocessing 단계입니다. Image processing 단계에서 중요시하게 생각한 것은 input image 들의 alignment입니다.
그 중 단연 첫 단계는 날 것의 이미지로부터 얼굴을 인식해내는 단계입니다. 현실 세계 속 조작되지 않은 이미지를 input 으로 받을 수 있기 위해서 필요한 단계입니다. 논문에서는 “A convolutional neural network cascade for face detection” 에서 구현한 Deep Convolutional Neural Network(DCNN) 이어붙인 모델의 얼굴 인식을 이용했습니다.

Test pipeline of facial detector [출처: A convolutional neural network cascade for face detection]
다음 단계는 제멋대로인 각각의 이미지들을 align하는 단계입니다. 이 과정은 이후 사용될 image combine을 비롯해 input image의 fomat을 통일하여 좋은 학습효과를 가져오기 위한 단계입니다. 논문에서는 130개의 얼굴 속 특징적인 지점(facial landmarks)를 찾아내어 이를 이용해 alignment를 진행한다고 합니다. Alignment를 통한 성능 개선은 논문 “Learning to Align from Scratch”에서 찾아볼 수 있습니다.

Sample images from LFW produced by different alignment algorithms [출처: Learning to Align from Scratch]
마지막 단계는 align된 이미지를 고정된 사이즈 100 * 100의 형태로 변환하고 회색조(grayscale)로 변환하는 과정입니다. 더불어 얼굴의 특정 영역(facial region)에 대한 선택적으로 이미지를 자르는 과정도 포함합니다. 이를 진행하는 이유는 본 포스트의 마지막에서 설명드리겠습니다. 이 과정을 마치면 input을 모델에 넣을 준비가 완료된 것입니다.
Phenotype extraction & Syndromes classification
논문에서 가장 도전적이었던 문제점 중 하나는 희귀 질환을 가진 환자들의 데이터의 수가 학습시키기에 부족했다는 점이었습니다. 이를 해결하기 위해서 논문에서는 두 단계로 학습을 진행시켰습니다.
첫 번째 단계는 baseline face representation을 위한 학습입니다. 이는 이미지 속에서 얼굴을 표현하는 단계입니다. 이전에 image preprocessing 에서도 진행한 단계이지만, 이 단계에서도 진행하는 것은 다음 단계에서 진행할 fine-tuning의 초기 값(initial weight)을 모델에 설정해두기 위함입니다.
다음 단계는 genetic syndrome classification을 위한 학습입니다. 이 단계에서는 이전 단계에서 구한 weight값을 업데이트하여 얼굴 정보 표현에 그쳤던 모델이 얼굴 정보 표현과 더불어 그 표현 속 유전성 질환 정보를 학습하기 위함입니다.
아래는 모델의 아키텍쳐에 대한 그림입니다.

The Deep Convolutional Neural Network architecture of DeepGestalt [출처: DeepGestalt-Identifying Rare Genetic Syndromes Using Deep Learning]
Choosing dataset
논문에서는 모델의 학습에 필요한 dataset으로 CASIA Web-Face dataset 과 Face2Gene phenotype dataset을 사용했습니다.
CASIA Web-Face dataset은 baseline face representation을 학습시키기 위한 용도로 사용되었고 10575명의 사람으로부터 얻어진 494414 images 를 포함하고 있습니다.
Face2Gene phenotype dataset은 genetic syndrome classification을 학습시키기 위한 용도로 사용되었고 2500개의 질환에 대한 수만개의 images 를 포함하고 있습니다.
Training
딥러닝에서 weight의 초기값 설정은 굉장히 중요한 일입니다. 초기값 설정에 따라 학습이 올바른 방향으로 갈 수도 있고, local minimum에 빠져서 학습에 실패할 수도 있습니다. 이러한 문제를 해결하기 위해서 사용하는 것이 weight initializer 입니다.
여러 번의 시도 끝에 논문에서는 baseline face representation 에서는 He Norma Initializer를, genetic syndrome classification에서는 Xavier normal initializer를 사용했습니다. 이는 가장 좋은 성능을 내는 initializer를 선택적으로 채용한 것입니다.
또한, 논문에서는 augmentation 을 사용하여 데이터의 개수를 늘리고 성능을 개선했습니다. 논문에서 조작한 것은 image의 회전, vertical/horizontal shift, zoom, shear transformation 정도입니다.
이 외에도 논문에서 언급한 것은 learning rate, epoch, momentum 등의 정보입니다. 이는 추후에 deep learning에 대한 개관에 대해서 설명할 때 모아서 진행하겠습니다.
지금은 데이터를 이용해 training을 할 때 본 논문에서 중요시 한 점이 weight initialization인 것과 augmentation 이라는 점만 짚고 넘어가겠습니다.
Evaluation
논문에서 모델의 성능을 측정하기 위해 사용한 것은 top-K-accuracy 입니다.
Top-K-accuracy는 sorted list형태로 가능성을 예측한 모델에서 K번째 안에 실제 값이 존재하는 경우를 나타내는 척도입니다.
즉, top-10-accuracy 의 경우, 전체 test 데이터들 중 가능성을 예측한 sorted list의 10번째 순서 안에 실제값이 존재할 경우의 수로 볼 수 있습니다.
Experiments and Results
본 논문에서는 크게 3가지의 형태로 실험을 진행했습니다. 그 중에서 논문에서 가장 중요하게 생각했던 multi-class Gestalt Model에 대해서만 언급하도록 하겠습니다.
(나머지 두 개는 각각 구현한 Gestalt Model이 binary classfication problem에도 잘 적용되는지와 genotype 예측에도 잘 적용되는지에 대한 실험이었습니다)
Multi-class Gestalt Model 은 논문에서 목표로 했던 바를 구현한 모델입니다. 다양한 질환을 가진 환자들이 섞여 있는 사진 속에서 각각의 사진에 대해서 사진 속 사람이 가지고 있는 희귀 질환의 가능성을 표현한 sorted list를 산출하는 것이 이 모델의 역할이었습니다.
216개의 유전성 희귀질환을 가진 26190개의 image를 통해 학습되고, 502개의 실제 환자의 image를 통해 테스트가 진행되었습니다.

DeepGestalt performance and permutation test result [출처: DeepGestalt-Identifying Rare Genetic Syndromes Using Deep Learning]
위 결과는 model의 accuracy 와 permutation test의 mean value 를 비교한 것입니다. 이 때 permutation test 의 경우 mean value를 test set으로 사용한 데이터의 label(여기서는 환자의 질환)을 마음대로 섞은 후 그 중 동일하게 맞을 경우의 수를 통해서 측정합니다.
당연스럽게도, 아무것도 하지 않은 채 추측한 것보다 논문에서 제시한 모델을 이용해 학습하여 산출한 결과 값이 더 높은 정확성을 가지고 의미있는 값을 나타내는 것을 알 수 있습니다.

Reason why the paper used cropped images and combine them [출처: DeepGestalt-Identifying Rare Genetic Syndromes Using Deep Learning]
앞서 말씀드린 내용 중에 짚고 넘어가지 않은 부분이 있습니다. 논문에서 image의 각각의 영역을 선택적으로 자르는 과정을 진행한다고 했었습니다. 이 부분에 대해서 논문에서 각 영역 별로의 학습 데이터로 추측한 accuracy 와 이를 합친 모델로 추측한 accuracy를 비교한 결과를 제공했습니다.
표를 보시면 Full Face 로 추측한 것보다 Aggregated model 로 추측한 것이 더 높은 accuracy를 가짐을 알 수 있습니다. 이것이 논문에서 input image 를 넣을 때 굳이 과정을 더해가면서 image를 자르고 각각을 학습시킨 이유입니다.
Conclusion
이것으로 논문 “DeepGestalt-Identifying Rare Genetic Syndromes Using Deep Learning”의 내용을 간단하게 요약해보았습니다. 중간중간에 언급하지는 않았지만 논문에서 강조한 논문의 의의는 random하게 sampling 된 images 에 대해서 높은 수준의 accuracy 로 유전성 희귀 질환을 진단해낼 수 있다는 점이었습니다.
이러한 형태의 딥러닝이 미래 정밀 의학에서 중요한 역할을 차지할 날이 머지 않을 것 같다고 느껴집니다.
Get to know us better! Join our official channels below.
Telegram(EN) : t.me/Humanscape KakaoTalk(KR) : open.kakao.com/o/gqbUQEM Website : humanscape.io Medium : medium.com/humanscape-ico Facebook : www.facebook.com/humanscape Twitter : twitter.com/Humanscape_io Reddit : https://www.reddit.com/r/Humanscape_official Bitcointalk announcement : https://bit.ly/2rVsP4T Email : [email protected]